DNA에 정보를 저장할 수 있나요?
왜 얼굴은 사람마다 다를까? 생물체의 모든 모양을 결정하는 유전자는 DNA 속에 들어 있다. DNA 속의 정보가 다르기 때문에 사람마다 얼굴 모습도 다르다. 생화학 분야와 분자생물학의 많은 과학자들은 부모의 모습이 다음 세대에 완벽하게 복제되어 전달될 수 있는지 오랫동안 연구되어 왔다. 프랜시스 크릭(Francis Crick)과 제임스 왓슨(James Watson)이 1953년에 DNA의 이중 나선 구조를 밝히면서 DNA 연구는 급진전 되었다. 이후 DNA의 염기 서열이 후대로 전달되는 것이 밝혀졌다. DNA는 유전 정보의 저장과 복제가 가능하다는 측면에서 견고한 하드 디스크에 비유할 수 있다. DNA를 응용한 다양한 기술에 대해 살펴본다.

DNA의 기본 구조
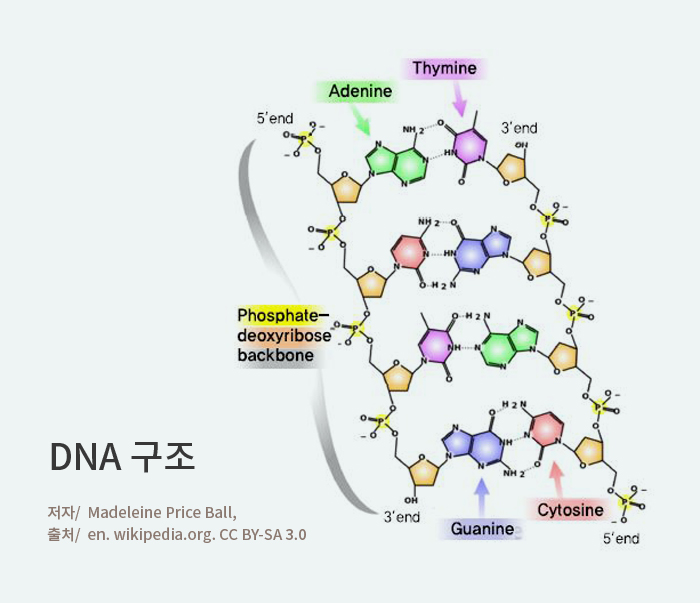
전 세계적으로 바이오 기술(BT: Bio Technology)의 주요 소재인 DNA(Deoxyribo Nucleic Acid, 디옥시리보핵산)을 정보기술(IT: Information Technology)과 나노 기술(NT: Nano Technology)에 적용하는 DNA 응용 기술 연구가 진행되고 있다. DNA의 기본 단위는 염기(Base), 5개의 탄소로 구성된 디옥시리보스(Deoxyrilbose, 갈색 오각형) 그리고 인산기(P: Phosphate, 노란색)가 결합된 뉴클레오타이드(Nucleotide)이다. DNA를 구성하는 염기에는 아데닌(A: Adenine), 티민(T: Thymine), 시토신(C: Cytosine) 그리고 구아닌(G: Guanine) 이렇게 4종류가 있기 때문에 뉴클레오타이드도 4종류이다.
2개의 수소 결합으로 A-T가 짝을 이루고, 3개의 수소 결합으로 G-C가 쌍을 이루며 존재하는데, 이러한 결합이 DNA의 상보성이다. 이는 DNA의 정보를 복제하고 저장하는 기능을 수행한다. DNA를 구성하는 4종류의 염기(A, T, C, G)는 배열되는 서열에 따라 유전 정보가 결정되며, 염기가 배열된 순서에 따라 단백질을 만드는 아미노산 순서도 결정된다.
우리 몸을 비롯한 모든 생명체는 다양한 세포로 이루어져 있다. 피부와 뼈, 혈액 등을 구성하는 각각의 세포는 각 세포마다 독특하고 다양한 단백질을 생성하며 생명을 유지한다. 이렇게 다양한 단백질은 20가지의 아미노산(Amino Acid)으로 구성된다. 각각의 단백질의 기능은 아미노산들의 배열 순서에 따라 결정되며, DNA 염기 서열에 의해 결정된다. DNA로부터 단백질을 생성하는 것은 복제(Replication), 전사(Transcription), 번역(Translation)의 세가지 순서로 이루어진다. 복제 과정에서 핵 속의 DNA 이중나선의 일부분이 열리고, 전사 과정에서는 DNA의 유전 정보를 읽은 뒤 유연하고 활용하기 쉬운 전령 RNA를 만든다. 번역 과정에서는 핵공을 통과해 핵 밖으로 나온 전령 RNA가 단백질 합성 기계인 리보솜(Ribosome)에 붙고, 리보솜은 전령 RNA의 코돈(Codon)을 바탕으로 아미노산 중합체인 단백질을 만든다. 운반 RNA(rRNA: transfer RNA)는 아미노산을 리보솜으로 운반한다.
DNA에 하드디스크와 같이 정보를 저장한다
데이터베이스(DB: DataBase) 혹은 비정형의 데이터로부터 가치 있는 정보를 추출하고 결과를 분석하는 기술인 빅데이터(Big Data) 시대를 맞이하였다. 의료, 과학 그리고 경제 분야 등 다양한 분야에서 중요성이 부각되고 있는 인공지능도 기본적으로 이러한 방대한 빅 데이터를 이용한다. 디지털 저장 장치는 쉽게 기록하고 저장할 수 있다는 장점이 있지만, 저장 수명이 짧은 게 단점이다. 이론적으로 DVD나 SSD(Solid State Drive) 같은 저장 장치는 최대 100년을 사용할 수 있지만, 현실적으로는 물리적 특성 때문에 이보다 훨씬 수명이 짧다. 또, 데이터센터의 운용과 유지비용에도 문제점이 있다. 데이터센터를 24시간 운영해야 하는 상황에서 저장 장치에서 발생하는 열을 식히기 위한 냉방 비용 및 저장 장치의 비용이 많이 소모된다. 이러한 상황에서 반영구적이고, 데이터를 저장하고 보관하는데 전기료와 같은 유지비용이 적게 드는 DNA를 활용한 저장 방법이 주목을 받고 있다.
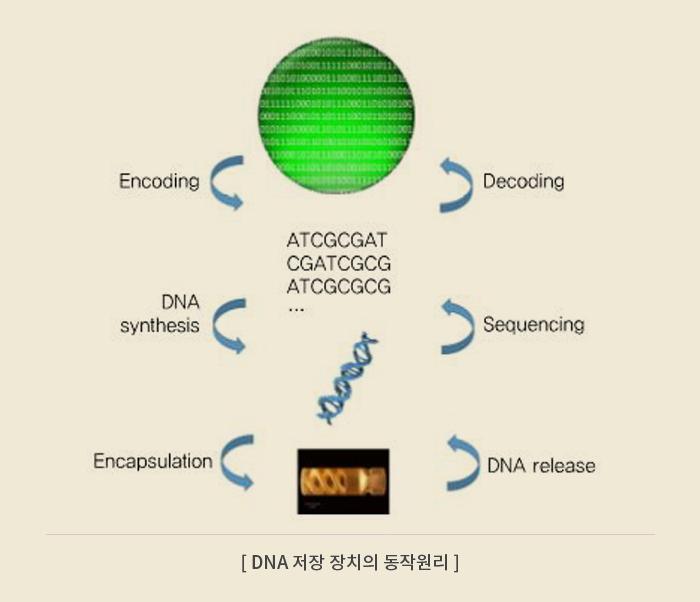
DNA 저장 장치의 또 다른 장점은 대용량의 데이터를 적은 양의 DNA로 저장할 수 있다는 것이다. 또한, DNA 저장 장치가 주목을 받는 이유 중 하나는 염기 서열을 인공적으로 합성하는 DNA 합성(DNA synthesis) 기술과 DNA 염기 서열을 읽는 시퀀싱(Sequencing) 기술이 비약적으로 발전했기 때문이다.
DNA 합성 기술은 데이터를 쓰는 기술이며, 시퀀싱 기술은 데이터를 읽는 기술이다. DNA는 네 가지 염기 서열을 이용해 0과 1로 이루어진 디지털 정보를 DNA 염기 서열로 인코딩한 뒤 캡슐화 하여 데이터를 저장한다. DNA 저장 장치에 대한 R&D는 해외에서 활발히 진행 중이다. 미국에서는 책 1권을 DNA 1mg에 저장한 후 3개월 후에 DNA 메모리에서 책 내용을 복원하는 데 성공하였으며, 영국에서는 100g 미만의 USB 3.0의 인터페이스를 가진 USB 크기의 DNA sequencer인 MinION을 개발하였다.
DNA 컴퓨터
DNA는 저장 장치 이외에도 DNA 컴퓨터 R&D가 연구되고 있다. 현재 사용하고 있는 컴퓨터는 CPU, 메모리 그리고 프로그램으로 구성된 폰노이만 구조이다. 이러한 방식의 컴퓨터는 프로그램에 의해 계산을 순차적으로 수행하며, 주소에 의해 메모리에 순차적으로 접근해서 데이터를 읽고 저장하는데, 고속 컴퓨터에서 이러한 순차적 처리 방법 때문에 병목 현상이 발생하는 문제가 있다. 반면, 인간의 뇌는 많은 뉴런이 동시에 연산에 참여하는 병렬적인 계산 방식을 사용하며, 주소 기반이 아닌 연산 능력을 이용하여 문제를 해결할 뿐만 아니라, 에너지 소모도 거의 없다. 이러한 인간의 뇌의 동작 방식을 CPU에 적용한 것이 뉴로모픽(Nuromorphic)칩이다. 인텔과 퀼컴에서 뉴로모픽칩 설계와 프로세서를 발표했지만, 뉴로모픽칩 또한 실리콘을 사용하기 때문에, 집적화에 따른 발열과 양자역학의 불확정성 원리에 의한 누전 등 많은 문제점을 내포하고 있다. 따라서 1994년 컴퓨터 공학자 아들만(Leonardo M. Adleman)에 의해 시작된 유전적 정보를 담고 있는 DNA를 컴퓨터에 적용하는 연구를 통해 실리콘 컴퓨터 시대 이후를 대비하기 위한 연구가 지속되고 있다.
DNA 컴퓨터는 사칙연산 연산자를 사용하는 기존의 컴퓨터와 달리 생체 분자를 이용한다. 해외에서는 Stanford 대학의 드류 앤디(Drew Endy)가 이끄는 Bioengineer 팀이 트랜스크립터(Transcriptor) 개발을 발표했다. DNA로 구성된 실리콘으로 비유하자면 트랜지스터 같은 장치이며, 기존의 컴퓨터 사칙연산과 같은 AND, OR, NOR 그리고 XNOR 등과 같은 연산 수행이 가능한 소자이다. 국내에서는 서울대학교 컴퓨터공학부 장병탁 교수가 DNA 컴퓨터를 만들어 인공지능의 성능을 향상하기 위한 인공지능 학습에 활용하고 있다. 아직 실리콘 기반의 컴퓨터를 대체할 만한 가시적인 성과가 미흡하지만, 계속해서 DNA 컴퓨터와 관련한 많은 연구가 진행되고 있으므로, 실리콘 이후 시대를 위한 DNA 컴퓨터가 상용화 될 것으로 예상한다.
DNA 나노 기술에 대한 지속적인 관심과 투자, 연구 필요
DNA 나노 기술은 4종류의 염기쌍들이 이중 혹은 삼중 수소 결합을 통해 안정적인 이중 나선 구조를 가지는 DNA 특성을 이용하여 나노미터 단위의 다양한 나노구조물을 만드는 기술이다. 즉, DNA를 단순히 유전정보의 저장 또는 전달 매체로 보지 않고 원하는 나노구조물을 만들기 위한 하나의 재료로 간주한 것이다.
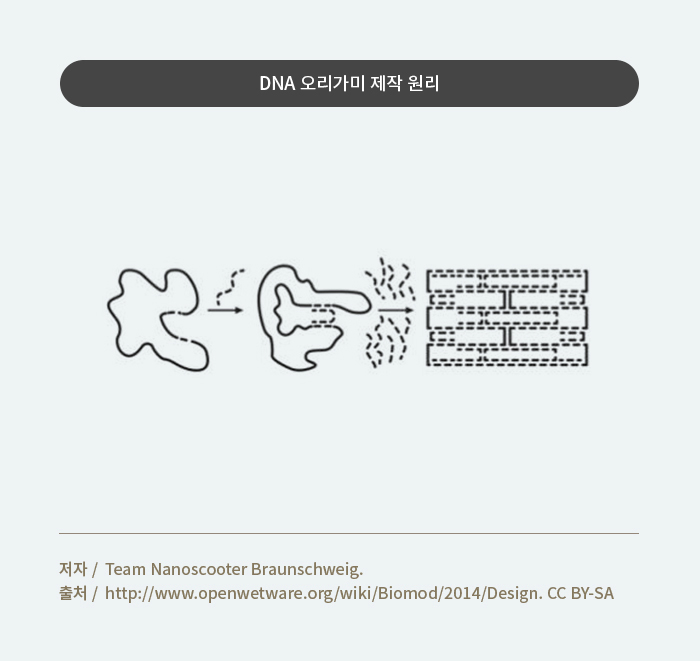
DNA를 이용하여 나노 단위의 구조물을 제작하는 방법 중에서 2006년 캘리포니아 공과대학교(Caltech: California Institute of Technology)의 폴 로데문드(Paul Rothemund) 교수의 DNA 오리가미(origami) 나노 기술이 있다. 오리가미는 일본말로 종이접기를 뜻한다. DNA 오리가미 나노 기술은 스캐폴드(Scaffold)와 스테이플(Staple)을 이용하여 열풀림(Thermal annealing)기법을 통해 나노구조물을 만들었다. 열풀림 방법을 사용하면 스테이플들이 스캐폴드의 지정된 위치에 염기의 상보적 서열 결합을 하는 자기조립(Self-assembly) 특성을 이용하여 DNA 나노구조물을 만들게 된다. 이러한 DNA 오리가미 기술은 타 나노공정 대비 비교적 단순하다는 장점이 있다. 이렇게 만들어진 2차원 나노구조물을 컴퓨터 프로그램을 이용하여 3차원 나노구조물로도 만들 수 있다. 컴퓨터 프로그램을 사용하는 이유는 사람이 일일이 7천개 이상의 염기 서열을 갖는 스캐필드를 실수 없이 정확히 설계하는데는 한계가 있기 때문이다. 이때 사용하는 컴퓨터 프로그램 중에 가장 대표적으로 사용되는 프로그램은 하버드 대학 윌리엄 쉬(William Shih) 교수가 개발한 3차원 랜더링 기능을 포함한 Cadnano이다. 나노스쿠터 팀은 DNA 오리가미 기술과 8,064개의 뉴클레오타이드로 구성된 스캐폴드를 사용하여 3차원 나노스쿠터를 제작하였다. DNA를 이용한 다른 분야는 DNA 나노튜브 하이브리드이며, 이러한 새로운 방법을 이용함으로써 나노 튜브 및 화학적 센서 등 많은 분야에 도움이 될 것으로 기대된다.

※ 논문 다운받기(논문 저자 : 이재호, 김도영, 박문호, 박윤옥 씨앗기술연구실 책임연구원 / 최윤호 씨앗기술연구실 선임연구원)
- 본 글은 전자통신동향분석 논문을 재구성하여 작성했습니다. 자세한 내용은 논문을 확인해주시기 바랍니다. -